# 分類準確性不夠:可以使用更多表現測量
> 原文: [https://machinelearningmastery.com/classification-accuracy-is-not-enough-more-performance-measures-you-can-use/](https://machinelearningmastery.com/classification-accuracy-is-not-enough-more-performance-measures-you-can-use/)
當您為分類問題構建模型時,您幾乎總是希望將該模型的準確性視為所有預測所做的正確預測的數量。
這是分類準確性。
在之前的文章中,我們研究了[評估模型](http://machinelearningmastery.com/how-to-choose-the-right-test-options-when-evaluating-machine-learning-algorithms/ "How To Choose The Right Test Options When Evaluating Machine Learning Algorithms")的穩健性,使用交叉驗證和多重交叉驗證來預測未見數據,其中我們使用了分類準確度和平均分類準確度。
一旦你擁有一個你認為可以做出強有力預測的模型,你需要確定它是否足以解決你的問題。單獨的分類準確性通常不足以做出此決定。
[](https://3qeqpr26caki16dnhd19sv6by6v-wpengine.netdna-ssl.com/wp-content/uploads/2014/03/classification-accuracy.jpg)
分類準確度
攝影:Nina Matthews攝影,保留一些權利
在這篇文章中,我們將介紹Precision和Recall表現度量,您可以使用它們來評估模型的二元分類問題。
## 乳腺癌復發
[乳腺癌數據集](http://archive.ics.uci.edu/ml/datasets/Breast+Cancer)是標準的機器學習數據集。它包含9個屬性,描述了286名患有乳腺癌并且在乳腺癌中存活并且在5年內是否復發的女性。
這是一個二元分類問題。在286名女性中,201名患者未復發乳腺癌,剩下的85名女性患乳腺癌。
我認為對于這個問題,假陰性可能比誤報更糟糕。你同意嗎?更詳細的篩查可以清除誤報,但是假陰性被送回家并丟失以進行后續評估。
## 分類準確性
[分類精度](http://en.wikipedia.org/wiki/Accuracy_and_precision)是我們的出發點。它是正確預測的數量除以預測的總數,乘以100將其變為百分比。
### 一切都沒有復發
僅預測不會復發乳腺癌的模型將達到(201/286)* 100或70.28%的準確度。我們稱之為“所有不復發”。這是一個高精度,但一個可怕的模型。如果它被單獨用于決策支持以告知醫生(不可能,但一起玩),它會將85名婦女誤認為他們的乳腺癌不會再發生(高假陰性)。
### 所有復發
僅預測乳腺癌復發的模型將達到(85/286)* 100或29.72%的準確度。我們稱之為“所有復發”。這個模型具有可怕的準確性,并且會讓201名女性認為乳腺癌復發,但實際上沒有(高假陽性)。
### 大車
CART或[分類和回歸樹](http://en.wikipedia.org/wiki/Predictive_analytics#Classification_and_regression_trees)是一種功能強大但簡單的決策樹算法。在這個問題上,CART可以達到69.23%的準確率。這低于我們的“All No Recurrence”模型,但這個模型更有價值嗎?
我們可以看出,單獨的分類準確性不足以為此問題選擇模型。
## 混亂矩陣
用于呈現分類器的預測結果的清晰且明確的方式是使用[混淆矩陣](http://en.wikipedia.org/wiki/Table_of_confusion#Table_of_confusion)(也稱為[列聯表](http://en.wikipedia.org/wiki/Contingency_table))。
對于二元分類問題,該表有2行2列。頂部是觀察到的類標簽,而旁邊是預測的類標簽。每個單元格包含落入該單元格的分類器所做的預測數量。
[](https://3qeqpr26caki16dnhd19sv6by6v-wpengine.netdna-ssl.com/wp-content/uploads/2014/03/truth_table.png)
真相表混淆矩陣
在這種情況下,一個完美的分類器將正確預測201沒有復發和85復發,這將進入左上角的細胞沒有復發/沒有復發(真陰性)和右下角細胞復發/復發(真陽性)。
不正確的預測顯然會分解為另外兩個單元格。假陰性是分類器標記為不再發生的重復。我們沒有這些。假陽性不是分類器標記為重復的重復。
這是一個有用的表,它提供了數據中的類分布和分類器預測的類分布以及錯誤類型的細分。
### 所有無復發混淆矩陣
混淆矩陣突出顯示大量假陰性(85)。
[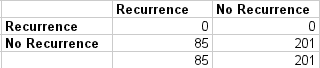](https://3qeqpr26caki16dnhd19sv6by6v-wpengine.netdna-ssl.com/wp-content/uploads/2014/03/no_recurrence_confusion_matrix.png)
所有無復發混淆矩陣
### 所有遞歸混淆矩陣
混淆矩陣突出了大量(201)的誤報。
[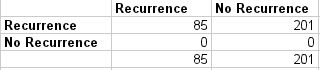](https://3qeqpr26caki16dnhd19sv6by6v-wpengine.netdna-ssl.com/wp-content/uploads/2014/03/recurrence_confusion_matrix.png)
所有遞歸混淆矩陣
### CART混淆矩陣
這看起來像一個更有價值的分類器,因為它正確地預測了10個重復事件以及188個沒有重復事件。該模型還顯示了適度數量的假陰性(75)和假陽性(13)。
[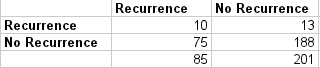](https://3qeqpr26caki16dnhd19sv6by6v-wpengine.netdna-ssl.com/wp-content/uploads/2014/03/cart_confusion_matrix.png)
CART混淆矩陣
## 準確性悖論
正如我們在這個例子中所看到的,準確性可能會產生誤導。有時可能需要選擇精度較低的模型,因為它對問題具有更強的預測能力。
例如,在存在大類不平衡的問題中,模型可以預測所有預測的多數類的值并實現高分類準確性,問題在于該模型在問題域中沒有用。正如我們在乳腺癌中看到的那樣。
這被稱為[準確性悖論](http://en.wikipedia.org/wiki/Accuracy_paradox)。對于類似的問題,需要這些額外的措施來評估分類器。
## 精確
[精度](http://en.wikipedia.org/wiki/Information_retrieval#Precision)是真陽性的數量除以真陽性和假陽性的數量。換句話說,它是正預測的數量除以預測的正類值的總數。它也被稱為[陽性預測值](http://en.wikipedia.org/wiki/Positive_predictive_value)(PPV)。
精度可以被認為是分類器精確度的度量。低精度也可以表示大量的誤報。
* All No Reurrence模型的精度為0 /(0 + 0)或不是數字,或0。
* All Recurrence模型的精度為85 /(85 + 201)或0.30。
* CART模型的精度為10 /(10 + 13)或0.43。
精度表明CART是一個更好的模型,即使它具有較低的準確度,所有重復發生比全無重復模型更有用。 All Reurrence模型和CART之間的精確度差異可以通過All Recurrence模型預測的大量誤報來解釋。
## 召回
[召回](http://en.wikipedia.org/wiki/Information_retrieval#Recall)是真陽性的數量除以真陽性的數量和假陰性的數量。換句話說,它是正預測的數量除以測試數據中的正類值的數量。它也稱為靈敏度或真陽性率。
召回可以被認為是分類器完整性的度量。低召回率表示許多假陰性。
* All No Recurrence模型的召回是0 /(0 + 85)或0。
* 召回全復發模型為85 /(85 + 0)或1。
* 召回CART為10 /(10 + 75)或0.12。
正如您所料,All Reurrence模型具有完美的回憶,因為它預測所有實例的“重復”。 CART的召回率低于All Recurrence模型的召回率。這可以通過CART模型預測的大量(75)假陰性來解釋。
## F1得分
[F1分數](http://en.wikipedia.org/wiki/F1_score)是2 *((精確*召回)/(精確+召回))。它也被稱為F分數或F量度。換句話說,F1分數表達了精確度和召回之間的平衡。
* 全無復發模型的F1為2 *((0 * 0)/ 0 + 0)或0。
* 全復發模型的F1為2 *((0.3 * 1)/0.3+1)或0.46。
* CART模型的F1為2 *((0.43 * 0.12)/0.43+0.12)或0.19。
如果我們希望基于精確度和召回之間的平衡來選擇模型,F1測量表明所有重復模型都是最佳模型,并且CART模型還沒有足夠的競爭力。
## 摘要
在這篇文章中,您了解了準確性悖論以及類別不平衡的問題,因為單獨的分類準確性無法被信任以選擇表現良好的模型。
通過示例,您了解了混淆矩陣,以此來描述未見數據集的預測中的錯誤細分。您了解了總結模型精度(準確性)和召回(完整性)的措施,以及F1分數中兩者之間平衡的描述。
- Machine Learning Mastery 應用機器學習教程
- 5競爭機器學習的好處
- 過度擬合的簡單直覺,或者為什么測試訓練數據是一個壞主意
- 特征選擇簡介
- 應用機器學習作為一個搜索問題的溫和介紹
- 為什么應用機器學習很難
- 為什么我的結果不如我想的那么好?你可能過度擬合了
- 用ROC曲線評估和比較分類器表現
- BigML評論:發現本機學習即服務平臺的聰明功能
- BigML教程:開發您的第一個決策樹并進行預測
- 構建生產機器學習基礎設施
- 分類準確性不夠:可以使用更多表現測量
- 一種預測模型的巧妙應用
- 機器學習項目中常見的陷阱
- 數據清理:將凌亂的數據轉換為整潔的數據
- 機器學習中的數據泄漏
- 數據,學習和建模
- 數據管理至關重要以及為什么需要認真對待它
- 將預測模型部署到生產中
- 參數和超參數之間有什么區別?
- 測試和驗證數據集之間有什么區別?
- 發現特征工程,如何設計特征以及如何獲得它
- 如何開始使用Kaggle
- 超越預測
- 如何在評估機器學習算法時選擇正確的測試選項
- 如何定義機器學習問題
- 如何評估機器學習算法
- 如何獲得基線結果及其重要性
- 如何充分利用機器學習數據
- 如何識別數據中的異常值
- 如何提高機器學習效果
- 如何在競爭機器學習中踢屁股
- 如何知道您的機器學習模型是否具有良好的表現
- 如何布局和管理您的機器學習項目
- 如何為機器學習準備數據
- 如何減少最終機器學習模型中的方差
- 如何使用機器學習結果
- 如何解決像數據科學家這樣的問題
- 通過數據預處理提高模型精度
- 處理機器學習的大數據文件的7種方法
- 建立機器學習系統的經驗教訓
- 如何使用機器學習清單可靠地獲得準確的預測(即使您是初學者)
- 機器學習模型運行期間要做什么
- 機器學習表現改進備忘單
- 來自世界級從業者的機器學習技巧:Phil Brierley
- 模型預測精度與機器學習中的解釋
- 競爭機器學習的模型選擇技巧
- 機器學習需要多少訓練數據?
- 如何系統地規劃和運行機器學習實驗
- 應用機器學習過程
- 默認情況下可重現的機器學習結果
- 10個實踐應用機器學習的標準數據集
- 簡單的三步法到最佳機器學習算法
- 打擊機器學習數據集中不平衡類的8種策略
- 模型表現不匹配問題(以及如何處理)
- 黑箱機器學習的誘惑陷阱
- 如何培養最終的機器學習模型
- 使用探索性數據分析了解您的問題并獲得更好的結果
- 什么是數據挖掘和KDD
- 為什么One-Hot在機器學習中編碼數據?
- 為什么你應該在你的機器學習問題上進行抽樣檢查算法
- 所以,你正在研究機器學習問題......
- Machine Learning Mastery Keras 深度學習教程
- Keras 中神經網絡模型的 5 步生命周期
- 在 Python 迷你課程中應用深度學習
- Keras 深度學習庫的二元分類教程
- 如何用 Keras 構建多層感知器神經網絡模型
- 如何在 Keras 中檢查深度學習模型
- 10 個用于 Amazon Web Services 深度學習的命令行秘籍
- 機器學習卷積神經網絡的速成課程
- 如何在 Python 中使用 Keras 進行深度學習的度量
- 深度學習書籍
- 深度學習課程
- 你所知道的深度學習是一種謊言
- 如何設置 Amazon AWS EC2 GPU 以訓練 Keras 深度學習模型(分步)
- 神經網絡中批量和迭代之間的區別是什么?
- 在 Keras 展示深度學習模型訓練歷史
- 基于 Keras 的深度學習模型中的dropout正則化
- 評估 Keras 中深度學習模型的表現
- 如何評價深度學習模型的技巧
- 小批量梯度下降的簡要介紹以及如何配置批量大小
- 在 Keras 中獲得深度學習幫助的 9 種方法
- 如何使用 Keras 在 Python 中網格搜索深度學習模型的超參數
- 用 Keras 在 Python 中使用卷積神經網絡進行手寫數字識別
- 如何用 Keras 進行預測
- 用 Keras 進行深度學習的圖像增強
- 8 個深度學習的鼓舞人心的應用
- Python 深度學習庫 Keras 簡介
- Python 深度學習庫 TensorFlow 簡介
- Python 深度學習庫 Theano 簡介
- 如何使用 Keras 函數式 API 進行深度學習
- Keras 深度學習庫的多類分類教程
- 多層感知器神經網絡速成課程
- 基于卷積神經網絡的 Keras 深度學習庫中的目標識別
- 流行的深度學習庫
- 用深度學習預測電影評論的情感
- Python 中的 Keras 深度學習庫的回歸教程
- 如何使用 Keras 獲得可重現的結果
- 如何在 Linux 服務器上運行深度學習實驗
- 保存并加載您的 Keras 深度學習模型
- 用 Keras 逐步開發 Python 中的第一個神經網絡
- 用 Keras 理解 Python 中的有狀態 LSTM 循環神經網絡
- 在 Python 中使用 Keras 深度學習模型和 Scikit-Learn
- 如何使用預訓練的 VGG 模型對照片中的物體進行分類
- 在 Python 和 Keras 中對深度學習模型使用學習率調度
- 如何在 Keras 中可視化深度學習神經網絡模型
- 什么是深度學習?
- 何時使用 MLP,CNN 和 RNN 神經網絡
- 為什么用隨機權重初始化神經網絡?
- Machine Learning Mastery 深度學習 NLP 教程
- 深度學習在自然語言處理中的 7 個應用
- 如何實現自然語言處理的波束搜索解碼器
- 深度學習文檔分類的最佳實踐
- 關于自然語言處理的熱門書籍
- 在 Python 中計算文本 BLEU 分數的溫和介紹
- 使用編碼器 - 解碼器模型的用于字幕生成的注入和合并架構
- 如何用 Python 清理機器學習的文本
- 如何配置神經機器翻譯的編碼器 - 解碼器模型
- 如何開始深度學習自然語言處理(7 天迷你課程)
- 自然語言處理的數據集
- 如何開發一種深度學習的詞袋模型來預測電影評論情感
- 深度學習字幕生成模型的溫和介紹
- 如何在 Keras 中定義神經機器翻譯的編碼器 - 解碼器序列 - 序列模型
- 如何利用小實驗在 Keras 中開發字幕生成模型
- 如何從頭開發深度學習圖片標題生成器
- 如何在 Keras 中開發基于字符的神經語言模型
- 如何開發用于情感分析的 N-gram 多通道卷積神經網絡
- 如何從零開始開發神經機器翻譯系統
- 如何在 Python 中用 Keras 開發基于單詞的神經語言模型
- 如何開發一種預測電影評論情感的詞嵌入模型
- 如何使用 Gensim 在 Python 中開發詞嵌入
- 用于文本摘要的編碼器 - 解碼器深度學習模型
- Keras 中文本摘要的編碼器 - 解碼器模型
- 用于神經機器翻譯的編碼器 - 解碼器循環神經網絡模型
- 淺談詞袋模型
- 文本摘要的溫和介紹
- 編碼器 - 解碼器循環神經網絡中的注意力如何工作
- 如何利用深度學習自動生成照片的文本描述
- 如何開發一個單詞級神經語言模型并用它來生成文本
- 淺談神經機器翻譯
- 什么是自然語言處理?
- 牛津自然語言處理深度學習課程
- 如何為機器翻譯準備法語到英語的數據集
- 如何為情感分析準備電影評論數據
- 如何為文本摘要準備新聞文章
- 如何準備照片標題數據集以訓練深度學習模型
- 如何使用 Keras 為深度學習準備文本數據
- 如何使用 scikit-learn 為機器學習準備文本數據
- 自然語言處理神經網絡模型入門
- 對自然語言處理的深度學習的承諾
- 在 Python 中用 Keras 進行 LSTM 循環神經網絡的序列分類
- 斯坦福自然語言處理深度學習課程評價
- 統計語言建模和神經語言模型的簡要介紹
- 使用 Keras 在 Python 中進行 LSTM 循環神經網絡的文本生成
- 淺談機器學習中的轉換
- 如何使用 Keras 將詞嵌入層用于深度學習
- 什么是用于文本的詞嵌入
- Machine Learning Mastery 深度學習時間序列教程
- 如何開發人類活動識別的一維卷積神經網絡模型
- 人類活動識別的深度學習模型
- 如何評估人類活動識別的機器學習算法
- 時間序列預測的多層感知器網絡探索性配置
- 比較經典和機器學習方法進行時間序列預測的結果
- 如何通過深度學習快速獲得時間序列預測的結果
- 如何利用 Python 處理序列預測問題中的缺失時間步長
- 如何建立預測大氣污染日的概率預測模型
- 如何開發一種熟練的機器學習時間序列預測模型
- 如何構建家庭用電自回歸預測模型
- 如何開發多步空氣污染時間序列預測的自回歸預測模型
- 如何制定多站點多元空氣污染時間序列預測的基線預測
- 如何開發時間序列預測的卷積神經網絡模型
- 如何開發卷積神經網絡用于多步時間序列預測
- 如何開發單變量時間序列預測的深度學習模型
- 如何開發 LSTM 模型用于家庭用電的多步時間序列預測
- 如何開發 LSTM 模型進行時間序列預測
- 如何開發多元多步空氣污染時間序列預測的機器學習模型
- 如何開發多層感知器模型進行時間序列預測
- 如何開發人類活動識別時間序列分類的 RNN 模型
- 如何開始深度學習的時間序列預測(7 天迷你課程)
- 如何網格搜索深度學習模型進行時間序列預測
- 如何對單變量時間序列預測的網格搜索樸素方法
- 如何在 Python 中搜索 SARIMA 模型超參數用于時間序列預測
- 如何在 Python 中進行時間序列預測的網格搜索三次指數平滑
- 一個標準的人類活動識別問題的溫和介紹
- 如何加載和探索家庭用電數據
- 如何加載,可視化和探索復雜的多變量多步時間序列預測數據集
- 如何從智能手機數據模擬人類活動
- 如何根據環境因素預測房間占用率
- 如何使用腦波預測人眼是開放還是閉合
- 如何在 Python 中擴展長短期內存網絡的數據
- 如何使用 TimeseriesGenerator 進行 Keras 中的時間序列預測
- 基于機器學習算法的室內運動時間序列分類
- 用于時間序列預測的狀態 LSTM 在線學習的不穩定性
- 用于罕見事件時間序列預測的 LSTM 模型體系結構
- 用于時間序列預測的 4 種通用機器學習數據變換
- Python 中長短期記憶網絡的多步時間序列預測
- 家庭用電機器學習的多步時間序列預測
- Keras 中 LSTM 的多變量時間序列預測
- 如何開發和評估樸素的家庭用電量預測方法
- 如何為長短期記憶網絡準備單變量時間序列數據
- 循環神經網絡在時間序列預測中的應用
- 如何在 Python 中使用差異變換刪除趨勢和季節性
- 如何在 LSTM 中種子狀態用于 Python 中的時間序列預測
- 使用 Python 進行時間序列預測的有狀態和無狀態 LSTM
- 長短時記憶網絡在時間序列預測中的適用性
- 時間序列預測問題的分類
- Python 中長短期記憶網絡的時間序列預測
- 基于 Keras 的 Python 中 LSTM 循環神經網絡的時間序列預測
- Keras 中深度學習的時間序列預測
- 如何用 Keras 調整 LSTM 超參數進行時間序列預測
- 如何在時間序列預測訓練期間更新 LSTM 網絡
- 如何使用 LSTM 網絡的 Dropout 進行時間序列預測
- 如何使用 LSTM 網絡中的特征進行時間序列預測
- 如何在 LSTM 網絡中使用時間序列進行時間序列預測
- 如何利用 LSTM 網絡進行權重正則化進行時間序列預測
- Machine Learning Mastery 線性代數教程
- 機器學習數學符號的基礎知識
- 用 NumPy 陣列輕松介紹廣播
- 如何從 Python 中的 Scratch 計算主成分分析(PCA)
- 用于編碼器審查的計算線性代數
- 10 機器學習中的線性代數示例
- 線性代數的溫和介紹
- 用 NumPy 輕松介紹 Python 中的 N 維數組
- 機器學習向量的溫和介紹
- 如何在 Python 中為機器學習索引,切片和重塑 NumPy 數組
- 機器學習的矩陣和矩陣算法簡介
- 溫和地介紹機器學習的特征分解,特征值和特征向量
- NumPy 對預期價值,方差和協方差的簡要介紹
- 機器學習矩陣分解的溫和介紹
- 用 NumPy 輕松介紹機器學習的張量
- 用于機器學習的線性代數中的矩陣類型簡介
- 用于機器學習的線性代數備忘單
- 線性代數的深度學習
- 用于機器學習的線性代數(7 天迷你課程)
- 機器學習的線性代數
- 機器學習矩陣運算的溫和介紹
- 線性代數評論沒有廢話指南
- 學習機器學習線性代數的主要資源
- 淺談機器學習的奇異值分解
- 如何用線性代數求解線性回歸
- 用于機器學習的稀疏矩陣的溫和介紹
- 機器學習中向量規范的溫和介紹
- 學習線性代數用于機器學習的 5 個理由
- Machine Learning Mastery LSTM 教程
- Keras中長短期記憶模型的5步生命周期
- 長短時記憶循環神經網絡的注意事項
- CNN長短期記憶網絡
- 逆向神經網絡中的深度學習速成課程
- 可變長度輸入序列的數據準備
- 如何用Keras開發用于Python序列分類的雙向LSTM
- 如何開發Keras序列到序列預測的編碼器 - 解碼器模型
- 如何診斷LSTM模型的過度擬合和欠擬合
- 如何開發一種編碼器 - 解碼器模型,注重Keras中的序列到序列預測
- 編碼器 - 解碼器長短期存儲器網絡
- 神經網絡中爆炸梯度的溫和介紹
- 對時間反向傳播的溫和介紹
- 生成長短期記憶網絡的溫和介紹
- 專家對長短期記憶網絡的簡要介紹
- 在序列預測問題上充分利用LSTM
- 編輯器 - 解碼器循環神經網絡全局注意的溫和介紹
- 如何利用長短時記憶循環神經網絡處理很長的序列
- 如何在Python中對一個熱編碼序列數據
- 如何使用編碼器 - 解碼器LSTM來回顯隨機整數序列
- 具有注意力的編碼器 - 解碼器RNN體系結構的實現模式
- 學習使用編碼器解碼器LSTM循環神經網絡添加數字
- 如何學習長短時記憶循環神經網絡回聲隨機整數
- 具有Keras的長短期記憶循環神經網絡的迷你課程
- LSTM自動編碼器的溫和介紹
- 如何用Keras中的長短期記憶模型進行預測
- 用Python中的長短期內存網絡演示內存
- 基于循環神經網絡的序列預測模型的簡要介紹
- 深度學習的循環神經網絡算法之旅
- 如何重塑Keras中長短期存儲網絡的輸入數據
- 了解Keras中LSTM的返回序列和返回狀態之間的差異
- RNN展開的溫和介紹
- 5學習LSTM循環神經網絡的簡單序列預測問題的例子
- 使用序列進行預測
- 堆疊長短期內存網絡
- 什么是教師強制循環神經網絡?
- 如何在Python中使用TimeDistributed Layer for Long Short-Term Memory Networks
- 如何準備Keras中截斷反向傳播的序列預測
- 如何在使用LSTM進行訓練和預測時使用不同的批量大小
- Machine Learning Mastery 機器學習算法教程
- 機器學習算法之旅
- 用于機器學習的裝袋和隨機森林集合算法
- 從頭開始實施機器學習算法的好處
- 更好的樸素貝葉斯:從樸素貝葉斯算法中獲取最多的12個技巧
- 機器學習的提升和AdaBoost
- 選擇機器學習算法:Microsoft Azure的經驗教訓
- 機器學習的分類和回歸樹
- 什么是機器學習中的混淆矩陣
- 如何使用Python從頭開始創建算法測試工具
- 通過創建機器學習算法的目標列表來控制
- 從頭開始停止編碼機器學習算法
- 在實現機器學習算法時,不要從開源代碼開始
- 不要使用隨機猜測作為基線分類器
- 淺談機器學習中的概念漂移
- 溫和介紹機器學習中的偏差 - 方差權衡
- 機器學習的梯度下降
- 機器學習算法如何工作(他們學習輸入到輸出的映射)
- 如何建立機器學習算法的直覺
- 如何實現機器學習算法
- 如何研究機器學習算法行為
- 如何學習機器學習算法
- 如何研究機器學習算法
- 如何研究機器學習算法
- 如何在Python中從頭開始實現反向傳播算法
- 如何用Python從頭開始實現Bagging
- 如何用Python從頭開始實現基線機器學習算法
- 如何在Python中從頭開始實現決策樹算法
- 如何用Python從頭開始實現學習向量量化
- 如何利用Python從頭開始隨機梯度下降實現線性回歸
- 如何利用Python從頭開始隨機梯度下降實現Logistic回歸
- 如何用Python從頭開始實現機器學習算法表現指標
- 如何在Python中從頭開始實現感知器算法
- 如何在Python中從零開始實現隨機森林
- 如何在Python中從頭開始實現重采樣方法
- 如何用Python從頭開始實現簡單線性回歸
- 如何用Python從頭開始實現堆棧泛化(Stacking)
- K-Nearest Neighbors for Machine Learning
- 學習機器學習的向量量化
- 機器學習的線性判別分析
- 機器學習的線性回歸
- 使用梯度下降進行機器學習的線性回歸教程
- 如何在Python中從頭開始加載機器學習數據
- 機器學習的Logistic回歸
- 機器學習的Logistic回歸教程
- 機器學習算法迷你課程
- 如何在Python中從頭開始實現樸素貝葉斯
- 樸素貝葉斯機器學習
- 樸素貝葉斯機器學習教程
- 機器學習算法的過擬合和欠擬合
- 參數化和非參數機器學習算法
- 理解任何機器學習算法的6個問題
- 在機器學習中擁抱隨機性
- 如何使用Python從頭開始擴展機器學習數據
- 機器學習的簡單線性回歸教程
- 有監督和無監督的機器學習算法
- 用于機器學習的支持向量機
- 在沒有數學背景的情況下理解機器學習算法的5種技術
- 最好的機器學習算法
- 教程從頭開始在Python中實現k-Nearest Neighbors
- 通過從零開始實現它們來理解機器學習算法(以及繞過壞代碼的策略)
- 使用隨機森林:在121個數據集上測試179個分類器
- 為什么從零開始實現機器學習算法
- Machine Learning Mastery 機器學習入門教程
- 機器學習入門的四個步驟:初學者入門與實踐的自上而下策略
- 你應該培養的 5 個機器學習領域
- 一種選擇機器學習算法的數據驅動方法
- 機器學習中的分析與數值解
- 應用機器學習是一種精英政治
- 機器學習的基本概念
- 如何成為數據科學家
- 初學者如何在機器學習中弄錯
- 機器學習的最佳編程語言
- 構建機器學習組合
- 機器學習中分類與回歸的區別
- 評估自己作為數據科學家并利用結果建立驚人的數據科學團隊
- 探索 Kaggle 大師的方法論和心態:對 Diogo Ferreira 的采訪
- 擴展機器學習工具并展示掌握
- 通過尋找地標開始機器學習
- 溫和地介紹預測建模
- 通過提供結果在機器學習中獲得夢想的工作
- 如何開始機器學習:自學藍圖
- 開始并在機器學習方面取得進展
- 應用機器學習的 Hello World
- 初學者如何使用小型項目開始機器學習并在 Kaggle 上進行競爭
- 我如何開始機器學習? (簡短版)
- 我是如何開始機器學習的
- 如何在機器學習中取得更好的成績
- 如何從在銀行工作到擔任 Target 的高級數據科學家
- 如何學習任何機器學習工具
- 使用小型目標項目深入了解機器學習工具
- 獲得付費申請機器學習
- 映射機器學習工具的景觀
- 機器學習開發環境
- 機器學習金錢
- 程序員的機器學習
- 機器學習很有意思
- 機器學習是 Kaggle 比賽
- 機器學習現在很受歡迎
- 機器學習掌握方法
- 機器學習很重要
- 機器學習 Q& A:概念漂移,更好的結果和學習更快
- 缺乏自學機器學習的路線圖
- 機器學習很重要
- 快速了解任何機器學習工具(即使您是初學者)
- 機器學習工具
- 找到你的機器學習部落
- 機器學習在一年
- 通過競爭一致的大師 Kaggle
- 5 程序員在機器學習中開始犯錯誤
- 哲學畢業生到機器學習從業者(Brian Thomas 采訪)
- 機器學習入門的實用建議
- 實用機器學習問題
- 使用來自 UCI 機器學習庫的數據集練習機器學習
- 使用秘籍的任何機器學習工具快速啟動
- 程序員可以進入機器學習
- 程序員應該進入機器學習
- 項目焦點:Shashank Singh 的人臉識別
- 項目焦點:使用 Mahout 和 Konstantin Slisenko 進行堆棧交換群集
- 機器學習自學指南
- 4 個自學機器學習項目
- álvaroLemos 如何在數據科學團隊中獲得機器學習實習
- 如何思考機器學習
- 現實世界機器學習問題之旅
- 有關機器學習的有用知識
- 如果我沒有學位怎么辦?
- 如果我不是一個優秀的程序員怎么辦?
- 如果我不擅長數學怎么辦?
- 為什么機器學習算法會處理以前從未見過的數據?
- 是什么阻礙了你的機器學習目標?
- 什么是機器學習?
- 機器學習適合哪里?
- 為什么要進入機器學習?
- 研究對您來說很重要的機器學習問題
- 你這樣做是錯的。為什么機器學習不必如此困難
- Machine Learning Mastery Sklearn 教程
- Scikit-Learn 的溫和介紹:Python 機器學習庫
- 使用 Python 管道和 scikit-learn 自動化機器學習工作流程
- 如何以及何時使用帶有 scikit-learn 的校準分類模型
- 如何比較 Python 中的機器學習算法與 scikit-learn
- 用于機器學習開發人員的 Python 崩潰課程
- 用 scikit-learn 在 Python 中集成機器學習算法
- 使用重采樣評估 Python 中機器學習算法的表現
- 使用 Scikit-Learn 在 Python 中進行特征選擇
- Python 中機器學習的特征選擇
- 如何使用 scikit-learn 在 Python 中生成測試數據集
- scikit-learn 中的機器學習算法秘籍
- 如何使用 Python 處理丟失的數據
- 如何開始使用 Python 進行機器學習
- 如何使用 Scikit-Learn 在 Python 中加載數據
- Python 中概率評分方法的簡要介紹
- 如何用 Scikit-Learn 調整算法參數
- 如何在 Mac OS X 上安裝 Python 3 環境以進行機器學習和深度學習
- 使用 scikit-learn 進行機器學習簡介
- 從 shell 到一本帶有 Fernando Perez 單一工具的書的 IPython
- 如何使用 Python 3 為機器學習開發創建 Linux 虛擬機
- 如何在 Python 中加載機器學習數據
- 您在 Python 中的第一個機器學習項目循序漸進
- 如何使用 scikit-learn 進行預測
- 用于評估 Python 中機器學習算法的度量標準
- 使用 Pandas 為 Python 中的機器學習準備數據
- 如何使用 Scikit-Learn 為 Python 機器學習準備數據
- 項目焦點:使用 Artem Yankov 在 Python 中進行事件推薦
- 用于機器學習的 Python 生態系統
- Python 是應用機器學習的成長平臺
- Python 機器學習書籍
- Python 機器學習迷你課程
- 使用 Pandas 快速和骯臟的數據分析
- 使用 Scikit-Learn 重新調整 Python 中的機器學習數據
- 如何以及何時使用 ROC 曲線和精確調用曲線進行 Python 分類
- 使用 scikit-learn 在 Python 中保存和加載機器學習模型
- scikit-learn Cookbook 書評
- 如何使用 Anaconda 為機器學習和深度學習設置 Python 環境
- 使用 scikit-learn 在 Python 中進行 Spot-Check 分類機器學習算法
- 如何在 Python 中開發可重復使用的抽樣檢查算法框架
- 使用 scikit-learn 在 Python 中進行 Spot-Check 回歸機器學習算法
- 使用 Python 中的描述性統計來了解您的機器學習數據
- 使用 OpenCV,Python 和模板匹配來播放“哪里是 Waldo?”
- 使用 Pandas 在 Python 中可視化機器學習數據
- Machine Learning Mastery 統計學教程
- 淺談計算正態匯總統計量
- 非參數統計的溫和介紹
- Python中常態測試的溫和介紹
- 淺談Bootstrap方法
- 淺談機器學習的中心極限定理
- 淺談機器學習中的大數定律
- 機器學習的所有統計數據
- 如何計算Python中機器學習結果的Bootstrap置信區間
- 淺談機器學習的Chi-Squared測試
- 機器學習的置信區間
- 隨機化在機器學習中解決混雜變量的作用
- 機器學習中的受控實驗
- 機器學習統計學速成班
- 統計假設檢驗的關鍵值以及如何在Python中計算它們
- 如何在機器學習中談論數據(統計學和計算機科學術語)
- Python中數據可視化方法的簡要介紹
- Python中效果大小度量的溫和介紹
- 估計隨機機器學習算法的實驗重復次數
- 機器學習評估統計的溫和介紹
- 如何計算Python中的非參數秩相關性
- 如何在Python中計算數據的5位數摘要
- 如何在Python中從頭開始編寫學生t檢驗
- 如何在Python中生成隨機數
- 如何轉換數據以更好地擬合正態分布
- 如何使用相關來理解變量之間的關系
- 如何使用統計信息識別數據中的異常值
- 用于Python機器學習的隨機數生成器簡介
- k-fold交叉驗證的溫和介紹
- 如何計算McNemar的比較兩種機器學習量詞的測試
- Python中非參數統計顯著性測試簡介
- 如何在Python中使用參數統計顯著性測試
- 機器學習的預測間隔
- 應用統計學與機器學習的密切關系
- 如何使用置信區間報告分類器表現
- 統計數據分布的簡要介紹
- 15 Python中的統計假設檢驗(備忘單)
- 統計假設檢驗的溫和介紹
- 10如何在機器學習項目中使用統計方法的示例
- Python中統計功效和功耗分析的簡要介紹
- 統計抽樣和重新抽樣的簡要介紹
- 比較機器學習算法的統計顯著性檢驗
- 機器學習中統計容差區間的溫和介紹
- 機器學習統計書籍
- 評估機器學習模型的統計數據
- 機器學習統計(7天迷你課程)
- 用于機器學習的簡明英語統計
- 如何使用統計顯著性檢驗來解釋機器學習結果
- 什么是統計(為什么它在機器學習中很重要)?
- Machine Learning Mastery 時間序列入門教程
- 如何在 Python 中為時間序列預測創建 ARIMA 模型
- 用 Python 進行時間序列預測的自回歸模型
- 如何回溯機器學習模型的時間序列預測
- Python 中基于時間序列數據的基本特征工程
- R 的時間序列預測熱門書籍
- 10 挑戰機器學習時間序列預測問題
- 如何將時間序列轉換為 Python 中的監督學習問題
- 如何將時間序列數據分解為趨勢和季節性
- 如何用 ARCH 和 GARCH 模擬波動率進行時間序列預測
- 如何將時間序列數據集與 Python 區分開來
- Python 中時間序列預測的指數平滑的溫和介紹
- 用 Python 進行時間序列預測的特征選擇
- 淺談自相關和部分自相關
- 時間序列預測的 Box-Jenkins 方法簡介
- 用 Python 簡要介紹時間序列的時間序列預測
- 如何使用 Python 網格搜索 ARIMA 模型超參數
- 如何在 Python 中加載和探索時間序列數據
- 如何使用 Python 對 ARIMA 模型進行手動預測
- 如何用 Python 進行時間序列預測的預測
- 如何使用 Python 中的 ARIMA 進行樣本外預測
- 如何利用 Python 模擬殘差錯誤來糾正時間序列預測
- 使用 Python 進行數據準備,特征工程和時間序列預測的移動平均平滑
- 多步時間序列預測的 4 種策略
- 如何在 Python 中規范化和標準化時間序列數據
- 如何利用 Python 進行時間序列預測的基線預測
- 如何使用 Python 對時間序列預測數據進行功率變換
- 用于時間序列預測的 Python 環境
- 如何重構時間序列預測問題
- 如何使用 Python 重新采樣和插值您的時間序列數據
- 用 Python 編寫 SARIMA 時間序列預測
- 如何在 Python 中保存 ARIMA 時間序列預測模型
- 使用 Python 進行季節性持久性預測
- 基于 ARIMA 的 Python 歷史規模敏感性預測技巧分析
- 簡單的時間序列預測模型進行測試,這樣你就不會欺騙自己
- 標準多變量,多步驟和多站點時間序列預測問題
- 如何使用 Python 檢查時間序列數據是否是固定的
- 使用 Python 進行時間序列數據可視化
- 7 個機器學習的時間序列數據集
- 時間序列預測案例研究與 Python:波士頓每月武裝搶劫案
- Python 的時間序列預測案例研究:巴爾的摩的年度用水量
- 使用 Python 進行時間序列預測研究:法國香檳的月銷售額
- 使用 Python 的置信區間理解時間序列預測不確定性
- 11 Python 中的經典時間序列預測方法(備忘單)
- 使用 Python 進行時間序列預測表現測量
- 使用 Python 7 天迷你課程進行時間序列預測
- 時間序列預測作為監督學習
- 什么是時間序列預測?
- 如何使用 Python 識別和刪除時間序列數據的季節性
- 如何在 Python 中使用和刪除時間序列數據中的趨勢信息
- 如何在 Python 中調整 ARIMA 參數
- 如何用 Python 可視化時間序列殘差預測錯誤
- 白噪聲時間序列與 Python
- 如何通過時間序列預測項目
- Machine Learning Mastery XGBoost 教程
- 通過在 Python 中使用 XGBoost 提前停止來避免過度擬合
- 如何在 Python 中調優 XGBoost 的多線程支持
- 如何配置梯度提升算法
- 在 Python 中使用 XGBoost 進行梯度提升的數據準備
- 如何使用 scikit-learn 在 Python 中開發您的第一個 XGBoost 模型
- 如何在 Python 中使用 XGBoost 評估梯度提升模型
- 在 Python 中使用 XGBoost 的特征重要性和特征選擇
- 淺談機器學習的梯度提升算法
- 應用機器學習的 XGBoost 簡介
- 如何在 macOS 上為 Python 安裝 XGBoost
- 如何在 Python 中使用 XGBoost 保存梯度提升模型
- 從梯度提升開始,比較 165 個數據集上的 13 種算法
- 在 Python 中使用 XGBoost 和 scikit-learn 進行隨機梯度提升
- 如何使用 Amazon Web Services 在云中訓練 XGBoost 模型
- 在 Python 中使用 XGBoost 調整梯度提升的學習率
- 如何在 Python 中使用 XGBoost 調整決策樹的數量和大小
- 如何在 Python 中使用 XGBoost 可視化梯度提升決策樹
- 在 Python 中開始使用 XGBoost 的 7 步迷你課程