## 16.1 統計建模過程
當我們想要使用我們的統計模型來檢驗一個科學假設時,我們通常要經歷一系列的步驟:
1. 指定您感興趣的問題
2. 識別或收集適當的數據
3. 準備數據進行分析
4. 確定合適的模型
5. 使模型適合數據
6. 批評模型以確保它適合
7. 檢驗假設和量化效應大小
讓我們來看一個真正的例子。2007 年,斯坦福大學的克里斯托弗·加德納和他的同事在美國醫學協會(HTG1)的雜志上發表了一項研究,題為“比較阿特金斯、Zone、Ornish 和學習飲食對超重絕經前婦女體重變化和相關危險因素的影響,A-toZ 減肥研究:一項隨機試驗”(Gardner 等人 2007 年)。
### 16.1.1 1:指定您感興趣的問題
據作者介紹,他們的研究目標是:
> 比較 4 種代表低到高碳水化合物攝入譜的減肥飲食對減肥和相關代謝變量的影響。
### 16.1.2 2:識別或收集適當的數據
為了回答他們的問題,研究人員隨機將 311 名超重/肥胖女性中的每一位分為四種不同的飲食(阿特金斯、Zone、Ornish 或 Learn),并隨著時間的推移跟蹤她們的體重和其他健康指標。
作者記錄了大量的變量,但對于主要的問題,讓我們關注一個變量:體重指數(bmi)。此外,由于我們的目標是測量體重指數的持續變化,我們將只觀察飲食開始后 12 個月的測量結果。
### 16.1.3 3:準備分析數據
A 到 Z 研究的實際數據并不公開,因此我們將使用他們論文中報告的摘要數據生成一些與他們研究中獲得的數據大致匹配的合成數據。
```r
# generate a dataset based on the results of Gardner et al. Table 3
set.seed(123456)
dietDf <-
data.frame(diet=c(rep('Atkins',77),
rep('Zone',79),
rep('LEARN',79),
rep('Ornish',76))) %>%
mutate(
BMIChange12Months=ifelse(diet=='Atkins',
rnorm(n=77,mean=-1.65,sd=2.54),
ifelse(diet=='Zone',
rnorm(n=79,mean=-0.53,sd=2.0),
ifelse(diet=='LEARN',
rnorm(n=79,mean=-0.92,sd=2.0),
rnorm(n=76,mean=-0.77,sd=2.14)))),
physicalActivity=ifelse(diet=='Atkins',
rnorm(n=77,mean=34,sd=6),
ifelse(diet=='Zone',
rnorm(n=79,mean=34,sd=6.0),
ifelse(diet=='LEARN',
rnorm(n=79,mean=34,sd=5.0),
rnorm(n=76,mean=35,sd=7) )))
)
summaryDf <-
dietDf %>%
group_by(diet) %>%
summarize(
n=n(),
meanBMIChange12Months=mean(BMIChange12Months),
varBMIChange12Months=var(BMIChange12Months)
) %>%
mutate(
crit_val_lower = qt(.05, n - 1),
crit_val_upper = qt(.95, n - 1),
ci.lower=meanBMIChange12Months+(sqrt(varBMIChange12Months)*crit_val_lower)/sqrt(n),
ci.upper=meanBMIChange12Months+(sqrt(varBMIChange12Months)*crit_val_upper)/sqrt(n)
)
summaryDf %>%
dplyr::select(-crit_val_lower,-crit_val_upper) %>%
pander()
```
<colgroup><col style="width: 10%"> <col style="width: 6%"> <col style="width: 28%"> <col style="width: 27%"> <col style="width: 13%"> <col style="width: 13%"></colgroup>
| 飲食 | N 號 | 平均質量變化 12 個月 | varbmichange12 個月 | CI.下 | CI.上部 |
| --- | --- | --- | --- | --- | --- |
| 阿特金斯 | 77 | -1.62 條 | 6.52 條 | -第 2.11 條 | -1.14 條 |
| 學習 | 79 | -0.85 分 | 3.77 條 | -1.21 條 | -0.49 分 |
| 尼什 | 76 | -0.69 分 | 5.36 條 | -1.13 條 | -0.25 分 |
| 區域 | 79 | -0.57 分 | 3.76 條 | -0.93 分 | -0.21 分 |
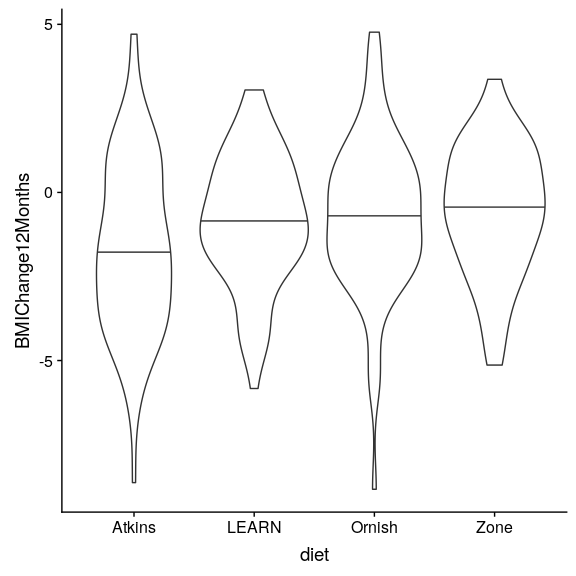
圖 16.1 每種情況下的小提琴圖,第 50 百分位(即中位數)顯示為每組的黑線。
現在我們有了數據,讓我們可視化它們,以確保沒有異常值。小提琴圖有助于觀察分布的形狀,如圖[16.1](#fig:AtoZBMIChangeDensity)所示。這些數據看起來相當合理——特別是,似乎沒有任何嚴重的異常值。然而,我們可以看到,分布似乎在方差上有點變化,阿特金斯和歐尼斯比其他分布顯示出更大的變異性。這意味著任何假設組間方差相等的分析都可能是不適當的。
### 16.1.4 4.確定合適的模型
為了確定適合我們分析的統計模型,我們需要問幾個問題。
* 什么樣的因變量?
* bmi:連續,大致正態分布
* 我們在比較什么?
* 四個飲食組的平均體重指數
* 方差分析是合適的
* 觀察是否獨立?
* 隨機分配和使用差分應確保獨立和相同分布(IID)誤差的假設是適當的。
### 16.1.5 5.使模型適合數據
讓我們做一個關于體重指數變化的方差分析來比較這四種飲食。事實證明,我們實際上不需要自己生成偽編碼變量;如果我們給`lm()`一個分類變量,它將自動為我們生成它們。
```r
# perform ANOVA and print result
lmResult <- lm(BMIChange12Months ~ diet, data = dietDf)
lmResult
```
```r
##
## Call:
## lm(formula = BMIChange12Months ~ diet, data = dietDf)
##
## Coefficients:
## (Intercept) dietLEARN dietOrnish dietZone
## -1.622 0.772 0.932 1.050
```
請注意,lm 會自動生成與四種飲食中的三種相對應的虛擬變量,使 atkins 飲食沒有虛擬變量。這意味著攔截模型阿特金斯飲食,其他三個變量模型的差異,這些飲食和阿特金斯飲食。
### 16.1.6 6.批評模型以確保它適合
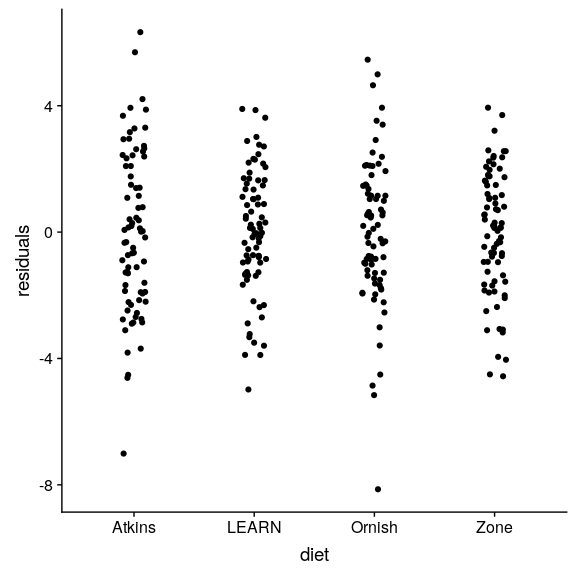
圖 16.2 分別繪制各組模型的殘差。
我們要做的第一件事就是批評這個模型,以確保它是適當的。我們可以做的一件事是從模型中觀察殘差。在圖[16.2](#fig:residPlot)中,我們將繪制按飲食分組的每個個體的殘差。我們已經把這些要點抖了抖,以便能看到所有的要點。不同條件下的殘差沒有明顯差異,這表明我們可以向前推進并解釋模型輸出。
### 16.1.7 7.檢驗假設和量化效應大小
首先,讓我們看一下方差分析的結果總結:
```r
# print summary of ANOVA statistics
summary(lmResult)
```
```r
##
## Call:
## lm(formula = BMIChange12Months ~ diet, data = dietDf)
##
## Residuals:
## Min 1Q Median 3Q Max
## -8.14 -1.37 0.07 1.50 6.33
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -1.622 0.251 -6.47 3.8e-10 ***
## dietLEARN 0.772 0.352 2.19 0.0292 *
## dietOrnish 0.932 0.356 2.62 0.0092 **
## dietZone 1.050 0.352 2.98 0.0031 **
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 2.2 on 307 degrees of freedom
## Multiple R-squared: 0.0338, Adjusted R-squared: 0.0243
## F-statistic: 3.58 on 3 and 307 DF, p-value: 0.0143
```
有意義的 F 檢驗表明,飲食之間有顯著的差異,但我們也應該注意,模型實際上并不能解釋數據的很大差異;R 平方值僅為 0.03,表明模型只占體重差異的百分之幾。t 損失。因此,我們不想曲解這個結果。
顯著的結果也不能告訴我們哪些飲食與其他飲食不同。我們可以通過使用`emmeans()`(“估計邊際平均值”)函數比較不同條件下的平均值來了解更多信息:
```r
# compute the differences between each of the means
leastsquare <- emmeans(lmResult,
pairwise ~ diet,
adjust="tukey")
# display the results by grouping using letters
CLD(leastsquare$emmeans,
alpha=.05,
Letters=letters)
```
```r
## diet emmean SE df lower.CL upper.CL .group
## Atkins -1.62 0.251 307 -2.11 -1.13 a
## LEARN -0.85 0.247 307 -1.34 -0.36 ab
## Ornish -0.69 0.252 307 -1.19 -0.19 b
## Zone -0.57 0.247 307 -1.06 -0.08 b
##
## Confidence level used: 0.95
## P value adjustment: tukey method for comparing a family of 4 estimates
## significance level used: alpha = 0.05
```
最右邊一列中的字母顯示了哪些組之間存在差異,使用的方法是根據正在執行的比較數量進行調整。這表明 Atkins 和 Learn 飲食沒有區別(因為它們共享字母 A),Learn、Ornish 和 Zone 飲食也沒有區別(因為它們共享字母 B),但 Atkins 飲食不同于 Ornish 和 Zone 飲食(因為它們不共享字母)。
#### 16.1.7.1 貝葉斯系數
假設我們希望有更好的方法來描述數據提供的證據數量。我們可以這樣做的一種方法是計算貝葉斯因子,我們可以通過擬合完整模型(包括飲食)和簡化模型(不包括飲食),然后比較它們的擬合度來實現這一點。對于簡化模型,我們只包含一個 1,它告訴擬合程序只適合一個截距。
```r
brmFullModel <- brm(BMIChange12Months ~ diet, data = dietDf,
save_all_pars = TRUE)
brmReducedModel <- brm(BMIChange12Months ~ 1, data = dietDf,
save_all_pars = TRUE)
```
```r
bayes_factor(brmFullModel,brmReducedModel)
```
```r
## Iteration: 1
## Iteration: 2
## Iteration: 3
## Iteration: 4
## Iteration: 1
## Iteration: 2
## Iteration: 3
## Iteration: 4
## Iteration: 5
```
```r
## Estimated Bayes factor in favor of bridge1 over bridge2: 99.86593
```
這表明,對于飲食之間的差異,有非常有力的證據(Bayes 因子接近 100)。
### 16.1.8 可能的混淆怎么辦?
如果我們更仔細地看加德的論文,我們會發現他們還報告了每組中有多少人被診斷為代謝綜合征(htg0),這是一種以高血壓、高血糖、腰部脂肪過多為特征的綜合征。膽固醇水平異常與心血管疾病風險增加有關。讓我們首先將這些數據添加到摘要數據框中:
```r
summaryDf <-
summaryDf %>%
mutate(
nMetSym=c(22,20,29,27),
nNoMetSym=n-nMetSym,
pMetSym=nMetSym/(nMetSym+nNoMetSym)
)
summaryDf %>%
dplyr::select(diet,n,pMetSym) %>%
pander()
```
<colgroup><col style="width: 12%"> <col style="width: 6%"> <col style="width: 12%"></colgroup>
| diet | n | PMETSYM 公司 |
| --- | --- | --- |
| Atkins | 77 | 0.29 分 |
| LEARN | 79 | 0.25 分 |
| Ornish | 76 | 0.38 分 |
| Zone | 79 | 0.34 分 |
從數據來看,似乎不同組之間的發病率略有不同,在華麗的飲食和區域性飲食中出現更多代謝綜合征病例——這正是結果較差的飲食。假設我們有興趣測試兩組之間的代謝綜合征發生率是否有顯著差異,因為這可能使我們擔心這些差異可能會影響飲食結果。
#### 16.1.8.1 確定適當的模型
* 什么樣的因變量?
* 比例
* 我們在比較什么?
* 四個飲食組與代謝綜合征的比例
* 擬合優度的卡方檢驗適用于零差假設。
讓我們使用`chisq.test()`函數計算該統計:
```r
chisq.test(summaryDf$nMetSym,summaryDf$nNoMetSym)
```
```r
##
## Pearson's Chi-squared test
##
## data: summaryDf$nMetSym and summaryDf$nNoMetSym
## X-squared = 10, df = 9, p-value = 0.2
```
這項測試表明,平均值之間沒有顯著差異。然而,它并沒有告訴我們有多確定我們是沒有區別的;記住,在 NHST 下,我們總是在假設無效為真的前提下工作,除非數據向我們展示足夠的證據,使我們拒絕這個無效假設。
如果我們想要量化支持或反對空值的證據呢?我們可以使用貝葉斯因子來實現這一點。
```r
bf <- contingencyTableBF(as.matrix(summaryDf[,9:10]),
sampleType = "indepMulti",
fixedMargin = "cols")
bf
```
```r
## Bayes factor analysis
## --------------
## [1] Non-indep. (a=1) : 0.058 ±0%
##
## Against denominator:
## Null, independence, a = 1
## ---
## Bayes factor type: BFcontingencyTable, independent multinomial
```
這表明,假設的概率比假設的概率高 0.058 倍,這意味著假設的概率比假設的概率高 1/0.058~17 倍。這是相當有力的證據,即使不是完全壓倒性的證據。
#### 16.1.8.2 解決模型中的混淆問題
我們通常使用隨機分配治療(如 Gardner 等人是的),因為我們認為,平均而言,它將阻止其他變量(我們稱之為 _ 混淆 _)對結果的影響。然而,正如我們在本例中看到的,在任何特定的研究中,可能會出現隨機發生的混淆。重要的是要確保條件之間的差異不是由于存在代謝綜合征的組之間的差異造成的,為此,我們可以在我們的統計模型中包括代謝綜合征變量。通過這樣做,任何與代謝綜合征相關的變異都將從飲食的影響中消除,這樣我們就可以更清楚地測量飲食的影響。
在這種情況下,我們沒有原始數據,因此無法直接測試它。如果我們有原始數據,我們可以在我們的模型中包括代謝綜合征變量,看看飲食和體重減輕之間的關系是否仍然有效;如果確實如此,那么我們可以更加確信,這種關系不是由代謝綜合征的任何差異所驅動的。
- 前言
- 0.1 本書為什么存在?
- 0.2 你不是統計學家-我們為什么要聽你的?
- 0.3 為什么是 R?
- 0.4 數據的黃金時代
- 0.5 開源書籍
- 0.6 確認
- 1 引言
- 1.1 什么是統計思維?
- 1.2 統計數據能為我們做什么?
- 1.3 統計學的基本概念
- 1.4 因果關系與統計
- 1.5 閱讀建議
- 2 處理數據
- 2.1 什么是數據?
- 2.2 測量尺度
- 2.3 什么是良好的測量?
- 2.4 閱讀建議
- 3 概率
- 3.1 什么是概率?
- 3.2 我們如何確定概率?
- 3.3 概率分布
- 3.4 條件概率
- 3.5 根據數據計算條件概率
- 3.6 獨立性
- 3.7 逆轉條件概率:貝葉斯規則
- 3.8 數據學習
- 3.9 優勢比
- 3.10 概率是什么意思?
- 3.11 閱讀建議
- 4 匯總數據
- 4.1 為什么要總結數據?
- 4.2 使用表格匯總數據
- 4.3 分布的理想化表示
- 4.4 閱讀建議
- 5 將模型擬合到數據
- 5.1 什么是模型?
- 5.2 統計建模:示例
- 5.3 什么使模型“良好”?
- 5.4 模型是否太好?
- 5.5 最簡單的模型:平均值
- 5.6 模式
- 5.7 變異性:平均值與數據的擬合程度如何?
- 5.8 使用模擬了解統計數據
- 5.9 Z 分數
- 6 數據可視化
- 6.1 數據可視化如何拯救生命
- 6.2 繪圖解剖
- 6.3 使用 ggplot 在 R 中繪制
- 6.4 良好可視化原則
- 6.5 最大化數據/墨水比
- 6.6 避免圖表垃圾
- 6.7 避免數據失真
- 6.8 謊言因素
- 6.9 記住人的局限性
- 6.10 其他因素的修正
- 6.11 建議閱讀和視頻
- 7 取樣
- 7.1 我們如何取樣?
- 7.2 采樣誤差
- 7.3 平均值的標準誤差
- 7.4 中心極限定理
- 7.5 置信區間
- 7.6 閱讀建議
- 8 重新采樣和模擬
- 8.1 蒙特卡羅模擬
- 8.2 統計的隨機性
- 8.3 生成隨機數
- 8.4 使用蒙特卡羅模擬
- 8.5 使用模擬統計:引導程序
- 8.6 閱讀建議
- 9 假設檢驗
- 9.1 無效假設統計檢驗(NHST)
- 9.2 無效假設統計檢驗:一個例子
- 9.3 無效假設檢驗過程
- 9.4 現代環境下的 NHST:多重測試
- 9.5 閱讀建議
- 10 置信區間、效應大小和統計功率
- 10.1 置信區間
- 10.2 效果大小
- 10.3 統計能力
- 10.4 閱讀建議
- 11 貝葉斯統計
- 11.1 生成模型
- 11.2 貝葉斯定理與逆推理
- 11.3 進行貝葉斯估計
- 11.4 估計后驗分布
- 11.5 選擇優先權
- 11.6 貝葉斯假設檢驗
- 11.7 閱讀建議
- 12 分類關系建模
- 12.1 示例:糖果顏色
- 12.2 皮爾遜卡方檢驗
- 12.3 應急表及雙向試驗
- 12.4 標準化殘差
- 12.5 優勢比
- 12.6 貝葉斯系數
- 12.7 超出 2 x 2 表的分類分析
- 12.8 注意辛普森悖論
- 13 建模持續關系
- 13.1 一個例子:仇恨犯罪和收入不平等
- 13.2 收入不平等是否與仇恨犯罪有關?
- 13.3 協方差和相關性
- 13.4 相關性和因果關系
- 13.5 閱讀建議
- 14 一般線性模型
- 14.1 線性回歸
- 14.2 安裝更復雜的模型
- 14.3 變量之間的相互作用
- 14.4“預測”的真正含義是什么?
- 14.5 閱讀建議
- 15 比較方法
- 15.1 學生 T 考試
- 15.2 t 檢驗作為線性模型
- 15.3 平均差的貝葉斯因子
- 15.4 配對 t 檢驗
- 15.5 比較兩種以上的方法
- 16 統計建模過程:一個實例
- 16.1 統計建模過程
- 17 做重復性研究
- 17.1 我們認為科學應該如何運作
- 17.2 科學(有時)是如何工作的
- 17.3 科學中的再現性危機
- 17.4 有問題的研究實踐
- 17.5 進行重復性研究
- 17.6 進行重復性數據分析
- 17.7 結論:提高科學水平
- 17.8 閱讀建議
- References
